 por Ignacio Wettling Carpio, Fonoaudiólogo, Magister en Trastornos del Lenguaje
por Ignacio Wettling Carpio, Fonoaudiólogo, Magister en Trastornos del Lenguaje
Introducción
Hace muy poco se difundió la noticia de la aparición de un Atlas del Cerebro humano, realizado por el Instituto Allen para Ciencias del Cerebro. En un principio no podría parecer ninguna novedad dado que ya existen otros como el creado por Paxino (Paxino, Assheuer y Mai, 2003), sin embargo lo significativo del hecho es que, junto con ser de acceso público, es el Atlas más completo sobre el cerebro hasta la fecha y permite acceder de forma gratuita a una amplia base de datos donde se puede cruzar información genómica, bioquímica y neuroanatómica (Figura 1). Como se logra esto? Vía web (funciona tanto en Windows como en Mac necesitando en ambos la última versión de Flash) y con un software especialmente diseñado, el Brain Explorer® 2, descargable desde la página del Instituto.
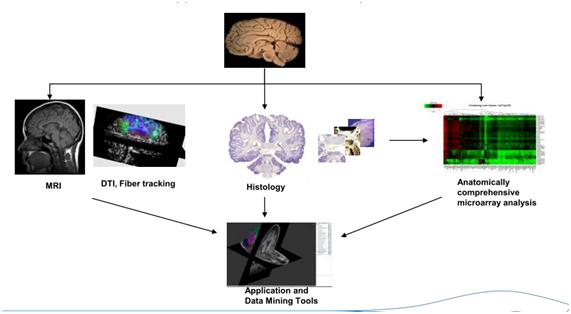 Figura 1 Esquema general del Atlas
Figura 1 Esquema general del Atlas
Como dato de interés, resta comentar que dicho Instituto el 2006 ya había hecho público anteriormente otros Atlas similares, el del cerebro del ratón, el de la médula espinal y el del cerebro del ratón en desarrollo, cuyos datos también están incluidos en parte en el Atlas del cerebro humano. Otro proyectos del Instituto en los cuales se basó la realización de este atlas, son (1) Medición de 100 Genes en la corteza Cortex; (2) Estudio sobre Esquizofrenia; (3) Estudio de Subcortex ; y (4) Atlas Transcripcional NIMH del desarrollo del cerebro humano, todos también disponibles desde el portal de Instituto.
Fundamentos
En propias palabras del Dr. Allan Jones, Ph.D en Neurociencias, académico de la UCLA y Director del Instituto Allen, el fin de dicho proyecto es entregar al mundo científico y de paso al público general, un recurso invaluable ya que tiene mapeado en su interior datos genéticos y bioquímicos de más de 1000 sitios específicos en el cerebro, lo que permitirá a investigadores de todo el mundo el poder comprobar hallazgos, plantear hipótesis y ahorrar recursos de manera considerable en la realización de estudios.
El Making Off
Se tomaron muestras postmortem de cerebros normales de individuos adultos (n= 2 – 6 por gen), los cuales se diseccionaron en pequeñas piezas de 3.5cm x 3.5cm. Estas muestras se sometieron a LCM (laser capture microdissection), revisadas por anatomistas expertos que ubicaron las zonas de interés y las llevaron a un modelo 3D. Luego esas muestras fueron analizadas utilizando DNA microarray (una técnica donde se comparan dos tejidos para detectar la expresión diferencial de genes y que permite la investigación del genoma completo, para más información sobre esta técnica pueden ir a http://www.quimicaviva.qb.fcen.uba.ar/V4n3/Barrero.htm para una buena descripción) y también utilizando la técnica hibridización in situ (se utiliza DNA o RNA complementario en un tejido bajo una tinción especial para localizar el gen, es una técnica para estudiar genes en particular). Con ellos se obtuvieron 50 millones de datos sobre la expresión génica, lo que se cruzó con la información anatómica obtenida previamente (Figura 2). Se debe agregar además que un hecho singular en la realización del atlas fue el descubrimiento que un 82% de todos los genes presentes en un humano se expresan en el cerebro. (Lo que llevo a que Jonah Lehrer, un columnista, asistente de Eric Kandel en la Universidad de Columbia y autor de artículos tales como How We Decide y Proust Was a Neuroscientist,, dijera la frase “the brain is one awesomely complicated piece of meat”!!, en una entrevista con el CEO del Instituto Allen acerca de este Atlas). Para más detalles respecto a las técnicas utilizadas en el análisis histológico, criterios utilizados en la delimitación de las zonas anatómicas, y los criterios de selección en la extracción de las muestras ir a http://human.brain-map.org/docs.html
 Y lo que queda podemos aprovecharlo en un buen asado…
Y lo que queda podemos aprovecharlo en un buen asado…
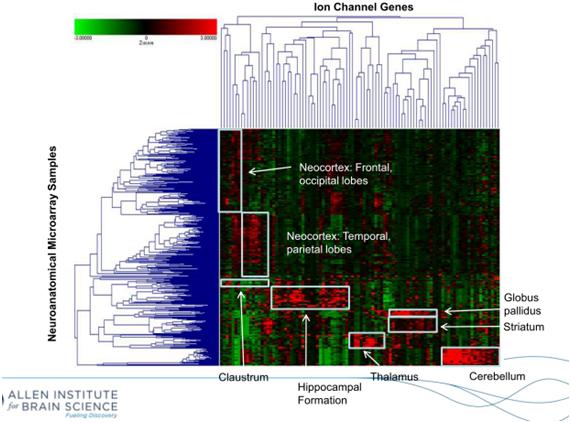 Figura 2: Estableciendo relaciones entre transcripción génica y estructuras cerebrales
Figura 2: Estableciendo relaciones entre transcripción génica y estructuras cerebrales
Revisión del Atlas
En el portal del Instituto, http://human.brain-map.org, al pinchar Human Brain, se accede a una barra con diversas opciones que dan cuenta de los diferentes aspectos del Atlas, los que se describirán a continuación
Microarray
Aquí es donde se realiza la búsqueda del gen (Gene search), que puede ser en forma aislada. Luego de esto aparece un Heat Map (Figura 3) donde se observan los datos obtenidos de la totalidad de donantes y las estructuras donde se expresa dicho gen (en el ejemplo utilice el FOXP2, que ha sido asociado a dificultades de habla y lenguaje).
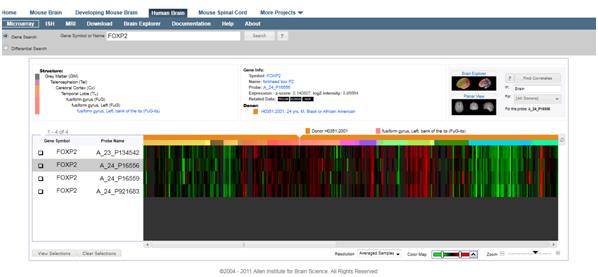 Figura 3: Parece chino mandarín…pero no, es peor
Figura 3: Parece chino mandarín…pero no, es peor
Luego de pinchar en algún punto del Heat Map que corresponderá a una zona anatómica específica donde se está expresando el gen. se observaran datos sobre el donante, las sondas (moléculas utilizadas en los análisis de expresión génica), el gen y da la posibilidad de ver anatómicamente las zonas de expresión en un gráfico planar (Figura 4). También da la posibilidad de verlos en un gráfico transplanar o en 3D mediante el Brain Explorer (debe descargarse antes)(Figura 5).
Dentro del microarray, existe otra opción además de la búsqueda simple del gen, y es el realizar una búsqueda diferencial (differential search) donde se comparan dos estructuras en base a la expresión génica presente en cada una de ellas, cuyos resultados son similares a los observados en la búsqueda simple, solo que esta vez se compararán ambas estructuras en un Heat Map.
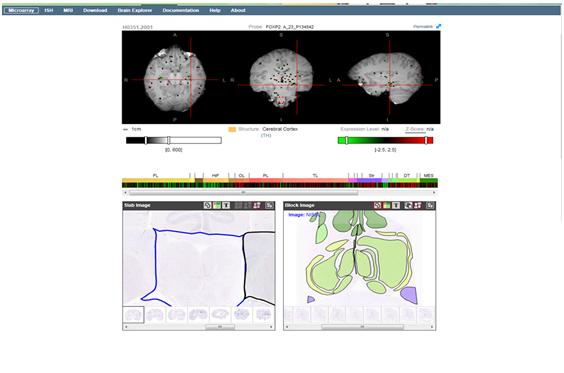
Figura 4: Y aquí es donde se expresa el gen
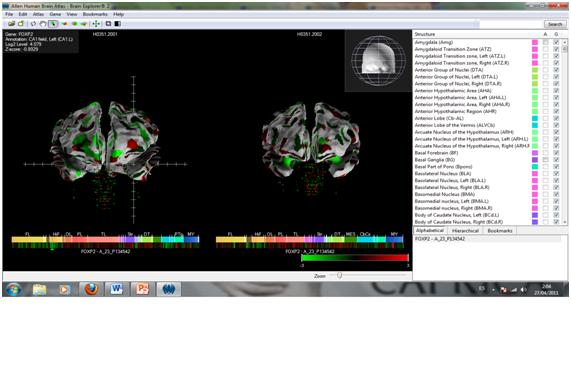
Figura 5: Acá en versión 3d gracias al Brain Explorer
Como vemos, es desde aquí es posible realizar el cruce de diversas modalidades de datos como son imágenes de resonancia magnética (MRI), expresión génica e histología (anatomía microscópica).
Hibridización in situ
Similar a lo observado en Microarray, pero además muestra temáticas de búsqueda como por ejemplo genes asociados a canales iónicos. Lo más interesante acá posiblemente sea la posibilidad de seleccionar los genes asociados a ciertas patologías como Autismo, Alzheimer, Esquizofrenia y otros (Figura 6)
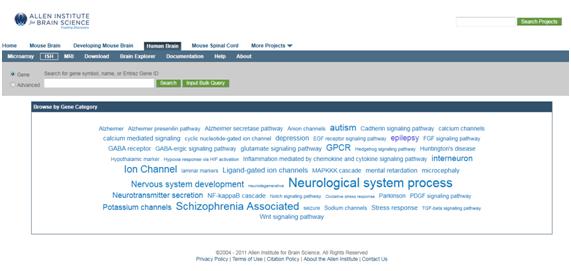 Figura 6 En búsqueda de los genes
Figura 6 En búsqueda de los genes
MRI
En esta sección se muestran imágenes de Resonancia Magnética, de algunos de los donantes (hay información contradictoria acá, puesto que se hablaba de 2 a 6 donantes distintos por gen, y en la página aparecen solo 4) que en este caso son 3 hombres y una mujer.
Otros elementos de la barra corresponden al sitio de descarga del Brain Explorer, la documentación, la ayuda y la descarga de las bases de datos.
Revision del Brain Explorer
Como ya se mencionó, el Brain Explorer 2 se encuentra descargable en forma gratuita desde http://human.brain-map.org/explorer.html, y permite visualizar las diferentes estructuras anatómicas del cerebro. En combinación con Microarray, puede visualizar la expresión génica en las diferentes zonas y por separado es un buen atlas 3D de las distintas estructuras cerebrales.
Se debe agregar que el software en si no es amigable (he aquí el infaltable manual http://community.brain-map.org/confluence/display/BrainExplorer/Home) aunque esto es esperable, dado que esta hecho principalmente para investigación de alto nivel, sin embargo su interfaz es intuitiva y permite la exploración de las diferentes estructuras de manera similar a como lo hace un programa de modelado 3D . Respecto a los requerimientos técnicos para que corra fluido, necesita un procesador Intel Core Duo or AMD de 1.8GHz, 1 Gb de RAM y una tarjeta de video de al menos 64 Mb.
Comentarios finales
Si bien el recurso es altamente específico y posiblemente muy poco utilizable para alguien sin mayores conocimientos sobre el área, los alcances que tendrá su uso en futuras investigaciones permitirá avances notables en el área de las Neurociencias y ramas afines. Esto se ve acrecentado debido a que la base de datos está siendo constantemente actualizada y por otro detalle interesante, pero lamentablemente no disponible para nuestro país, que es la realización de cursos de entrenamiento y capacitación para el uso del Atlas.
Entonces ya sabe, si quiere saber en qué lugar del cerebro se expresa un determinado gen, este Atlas (y un montón de conocimientos mas) puede ser de alta utilidad. Sin embargo, no olvidar que pese a que es un contenido de acceso libre, su uso en investigaciones debe ser correctamente citado (http://www.alleninstitute.org/citation_policy.htm). Finalmente se debe destacar el esfuerzo titánico de cada uno de los miembros Instituto Allen (Ver foto final) en la realización de lo que posiblemente sea uno de los aportes más significativos en los últimos tiempos al estudio del cerebro.
Fuentes: http://human.brain-map.org,
 Sigan asi muchachos!!
Sigan asi muchachos!!






3 comments
Hugo says:
Abr 29, 2011
Notable trabajo. Felicitaciones a Tercera Cultura y a Ignacio. Ojalá en Chile hubiese acceso a los medios que permitan aprovechar una herramienta como esta y sacarle el potencial más allá de ver ‘imágenes bonitas’.
Saludos!
Germán says:
Abr 29, 2011
la otra vez discutiamos la importancia y metodología de los microarreglos para detectar la expresion de genes, esta tecnología es un poco vieja y poco precisa, junto con ser meramente referencia. La idea principal es tener una gran libreria … de cDNA (secuencia de pares de bases que codifican exactamente para una proteína) de hebra simple, marcada fluorescentemente (reportero) y se mezclan con los mRNA o cDNA extraido del tejido (target), luego se mide la fluorescencia de lo que se unió, si fluoresce está y que tanto fluoresca puede indicar que tanto está.
Allen ya había hecho lo propio con su primer atlas, el del cerebro de rata, que ayudó a varias personas a establecer regiones donde se podrían expresar genes de interés y los llevó a mirar con detalle en esas zonas. A que voy, es que este e …s SOLO UNA REFERENCIA no un mapa como estamos acostumbrados, vale decir que la expresion de proteínas cambia según la edad y condiciones donde se encuentran los individuos, por lo que la presencia de una proteína en una región debe ser mirada con mas detalle y no es posible sacar conclusiones de este tipo de mapas, ayudan un montón y son super didacticos, pero no constituyen una aproximación precisa al analisis proteomico o genómico del cerebro.
Ricardo Martinez says:
Abr 29, 2011
Gracias Germán!